Vortexというpopulation simulatorで遊んでみた
×
[PR]上記の広告は3ヶ月以上新規記事投稿のないブログに表示されています。新しい記事を書く事で広告が消えます。
コメント
ただいまコメントを受けつけておりません。
Vortexというpopulation simulatorで遊んでみた
分子時計は現生人類の起源を探るのに強力な手法となっていますが、その年代推定は現状では非常に幅があります。mtDNAのハプログループL3とMおよびNが分岐した年代を出アフリカと考えますが、年代の幅は85000年~45000年前と非常に広くなっています。また常染色体での解析では、bottleneckが140000~80000年前とされるなど、mtDNAと一致しない結果も出ているようです。
mtDNA、Ychr、常染色体で分子時計にズレが出てくるのはどのような理由でしょうか。男性は多く移動し、極めて多数の子を残したりできるので遺伝子浮動の影響が大きい、とされることもあります。
ここでは、婚姻形態の影響について考えてみます。ある地方では、いくつか部族があって、部族同士で妻の交換はするが男は部族から動かない、としましょう。いわゆる嫁入り婚であり、わりとありそうな形です。この形態だと、常染色体(またはmtDNA)の遺伝子は移動する妻に乗って地方全体を動きます。しかしYchrはいつまでも部族の中から出ていきません。こうなると、常染色体(またはmtDNA)とYchrでeffective population sizeに大きな差が出てくるため、Ychrの方が遺伝子浮動の影響をより強く受けやすくなるような気がします。
この仮説を検証するのにシミュレーションという手法があります。例えばそれぞれ150人ずつの4つの村を想定します。男は村に残りますが、女は村を出て他村の男と結婚します。こうして何世代も計算して、最終的に遺伝子構成がどのような形になるかを見るのです。このようなシミュレーションで、遺伝子が婚姻形態でどのような影響をこうむるのか、実感できるはずです。
これは自分でプログラムを書く方法もありますが、簡単ではありません。さいわい今は交配と遺伝子のシミュレーションソフトが多数あります。
Computer simulations: tools for population and evolutionary genetics
Nature Reviews Genetics 13:110(2012)
に、40くらいのシミュレーターのレビューがありました。シミュレーターは大別して、現在の集団を設定して過去の系統関係をシミュレートするBackward simulatorと、最初の集団を設定してそれが時間経過でどのように変化するかを見るForward simulatorがあります。
このレビューにはシミュレーターの機能の一覧表があります。研究の目的にあわせてシミュレーターを選ぶためのチャートもあります。また、そもそもシミュレーション研究はどのような手順で進めるべきかという丁寧な指針も示されています。
今回の目的からすると、Forward simulatorが適当なようです。性別を限定した移動が可能なものが必要と思われます。正直UNIXはよくわからないし環境もないので避けたいし、できればコマンドラインよりしっかりGUIがあるほうがありがたいです。ただGUIのあるものは少なく、候補はだいぶ少なくなります。
今回はVortexというソフトを使ってみます。windows上で動くGUIのしっかりしたsimulatorで、かなり洗練されたソフトと思われます。Conservation breeding specialist groupのサイト(http://www.cbsg.org/)のScience-Based Toolsというページにあり、無料です。詳細なpdfのマニュアルもあります。そもそもは希少動物の保護のために、交配と個体数の予測をし、適切な介入を検討する目的で設計されたもののようです。かなり多機能なソフトなので、いろんなことができそうですが、設定もなかなかに大変そうです。遺伝子については常染色体とmtDNAを設定できるようです。Ychrとして解析することはできませんが、男女を入れ替えればmtDNAとしての結果を流用できそうです。
Simulation Inputから、多くのパラメーターを調節できます。
まず単純なモデルを目指します。
150人からなる4つの村で、女が動く、男が動く、どちらも動かない、を設定してみます。
Scenario Settingsで、まずScenario name、Number of iterations(シミュレーションの反復回数、まずは5~20回くらいから様子を見て、最終的な結果を出すときに数を増やす)、Number of years(timesteps)を設定します。時間はひとまず500年としてみます。
ここでNumber of populationsを4にすると、4つの村となります。
Species Descriptionでは、Inbreeding depressionをoffにして、近親交配による死亡率の上昇をないことにします。
Dispersalを設定して、他の村へ行く人数を決めます。
ここでは移動する年代を15歳のみとし、移動する性別を決めます。
そして、他村へ移動する割合を決めます。
おそらくこれで、15歳になったら、女性のほとんどが村を出て、他の3村に均等に移動する、ようになるはずです。
Don't allow dispersal into saturated populations、とはCarring Capacityがいっぱいになっている村へは移動しない、ということなので、これはチェックを外しておきます。
Reproductive SystemをLong-term monogamyとし、Age of first offspring females(males)を15、Maximum age of female(male) reproductionを35にします。男女ともに15歳で結婚し基本的にそのパートナーが変わらない、生殖期間は男女ともに15~35歳、となります
Reproductive Ratesから、女性の婚姻率を100%にし、1年に妊娠する確率を25%(つまり平均で4年に1回妊娠する)、子供はかならず1人(双子などは考慮しない)、とします。
Mortality Ratesは、生殖可能年齢までは各年齢ごとの死亡率を設定し、生殖可能年齢以後は毎年一定の死亡率となります。ただ、ヒトを想定した場合、各年齢ごとの死亡率を設定するのはかなり煩雑になります。なのでこのモデルでは0歳死亡率を設定し、1~14歳では死亡しないこととし、15歳以降死亡率を1%としています。
Catastrophes(天変地異)、Mate Monopolization(一夫多妻など独占の設定)は今回はなしとします。
Initial Population Size(最初の人口)は各村150人ずつです。Use stable age distributionにしておけば、死亡率から想定される人口構成を自動計算してくれます。これは死亡率の設定で人口に変な偏りが出ていないかどうかチェックするのにも使えます。
Carrying Capacityは各村150人とします。これは各村の人口的限界が150人ということであり、これを越えてしまった分は人口から排除されます(この排除の仕組みがまだよく理解できていません、、、)。
EVとはここまでも出てきていますが、environmental variableのことのようで、つまりその年の環境の厳しさを示す変数のようです。このような環境変動について今回はすべて無しとします。
Harvest、Supplementationは、それぞれ途中での人口の消失および追加を設定するものであり、今回は設定しません。
Geneticsは、肝心な部分ですが、とにかく他を先に設定してきちんと動くのを確かめてから設定します。(実はエラーが出たりしてまだ上手くここを扱えずにいます、、、、)
デフォルトでは、解析は常染色体の仮想locusひとつとmtDNAの仮想locusひとつについて行われ、開始時には全員のハプロタイプが異なるという設定になります。なので150人の村では、常染色体のとある仮想locusに(常染色体は2本なので)300のハプロタイプ、mtDNAのとある仮想locusに150のハプロタイプが存在する、というところから始まります。
このような初期設定は、Replace initial population with studbookや、Read initial allele frequenciesで設定するようです。
このようなモデルで一定の人口を保つのは難しいことです。調節のためには、Reproductive Systemで人口の多寡による婚姻率を調節する、GeneticsからBreed to maintain the population at Kをチェックする、という方法もありますが、今回は用いていません。
いずれにしても、Carrying Capacityによって、越えた分の人口を刈り取る必要がでてきます。ただ刈り取る人口は少なくしたいので、人口はCarrying Capacityがなければ微増する、という設定を目指します。
これは、Mortality Ratesで0歳時の死亡率を微調節することで設定しました。ツールボタンから「Det.」を押すと、理論上の人口増加率を見ることができます。実際にシミュレートするとランダムの影響で理論より増加率は低下してしまうので、少なくともDet.のrがマイナスにならないようにします。
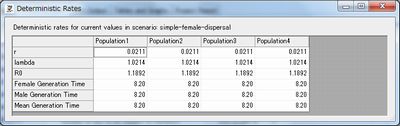
あとは少ない反復回数で何度かシミュレーションして調節します。今回0歳死亡率は50%としました。これが60%だと人口は微減し、55%くらいだと安定はしますがいったん人口が減ると回復できなくなりました。
シミュレーションを動かしてみます。すると、反復ごとの人口動態のグラフがリアルタイムで表示されます。
すべて終わると、結果はText Output, Tables and Graphsというタブから表示させることができます。
この設定では、100回反復して、村の絶滅は1件もありませんでした(設定を変えずにCarring Capacityを減らしたりすると結構すぐに絶滅します)。
N vs Yearで、年ごとの人口推移の平均が出ます。ほとんどCarrying Capacityいっぱいで一定になっているのがわかります。ただ、Dispersalありの方がほんのわずか人口が少なくなっているのが、シミュレーションのどこに原因があるのか検討が必要そうです。
常染色体仮想locusのAllele(ここでは ≒ハプロタイプ)は、初期設定では150人×4村×2本の染色体、で1200ありますが、急速に減少します。この減少度合いは、人口全体では全く差がありません。これは想定外の結果で、移動の全くない設定ではAlleleの減少はやや早いものと思っていました。検証が必要なところです。
一方村ひとつを取り出して見てみると、人口の移動がある方が、村としてのAlleleの数は保たれているのがわかります。
Text outputのOutput Summaryから、最終的なalleleの数およびmtDNAのハプロタイプの数を見ることができます。
今回のシミュレーションの結果です。
人口全体としては
女性のみが移動する場合
人口全体としては
女性のみが移動する場合
Across all years, prior to carrying capacity truncation,
mean growth rate (r) was 0.0059 (0.0000 SE, 0.0088 SD)
Final expected heterozygosity was 0.9670 (0.0005 SE; 0.0046 SD)
Final observed heterozygosity was 0.9713 (0.0009 SE; 0.0090 SD)
Final number of alleles was 56.5700 (0.4061 SE; 4.0608 SD)
Final number of mt haplotypes was 15.4800 (0.2218 SE; 2.2178 SD)
Mean Ne calculated from loss of heterozygosity (using T from 1st population): 932.55
男性のみが移動する場合
男性のみが移動する場合
Across all years, prior to carrying capacity truncation,
mean growth rate (r) was 0.0059 (0.0000 SE, 0.0087 SD)
Final expected heterozygosity was 0.9668 (0.0004 SE; 0.0045 SD)
Final observed heterozygosity was 0.9688 (0.0010 SE; 0.0097 SD)
Final number of alleles was 55.5300 (0.4721 SE; 4.7214 SD)
Final number of mt haplotypes was 15.0300 (0.2272 SE; 2.2717 SD)
Mean Ne calculated from loss of heterozygosity (using T from 1st population): 925.72
移動のまったくない場合
移動のまったくない場合
Across all years, prior to carrying capacity truncation,
mean growth rate (r) was 0.0059 (0.0000 SE, 0.0087 SD)
Final expected heterozygosity was 0.9694 (0.0004 SE; 0.0041 SD)
Final observed heterozygosity was 0.8885 (0.0022 SE; 0.0216 SD)
Final number of alleles was 58.8300 (0.4344 SE; 4.3439 SD)
Final number of mt haplotypes was 16.3200 (0.2304 SE; 2.3045 SD)
Mean Ne calculated from loss of heterozygosity (using T from 1st population): 1008.12
村1だけとしては、
女性のみが移動する場合
村1だけとしては、
女性のみが移動する場合
Across all years, prior to carrying capacity truncation,
mean growth rate (r) was 0.0055 (0.0001 SE, 0.0246 SD)
Final expected heterozygosity was 0.9626 (0.0006 SE; 0.0056 SD)
Final observed heterozygosity was 0.9717 (0.0015 SE; 0.0151 SD)
Final number of alleles was 47.71 (0.37 SE; 3.73 SD)
Final number of mt haplotypes was 14.20 (0.22 SE; 2.25 SD)
Mean Ne calculated from loss of heterozygosity in extant populations: 878.64
男性のみが移動する場合
このようなシミュレーションは強力なツールになりえますが、何かの結果がツールの問題なのか設定の問題なのか、真実なのか、検証が非常に難しいという印象です。男性のみが移動する場合
Across all years, prior to carrying capacity truncation,
mean growth rate (r) was 0.0056 (0.0001 SE, 0.0245 SD)
Final expected heterozygosity was 0.9625 (0.0006 SE; 0.0057 SD)
Final observed heterozygosity was 0.9691 (0.0016 SE; 0.0159 SD)
Final number of alleles was 47.20 (0.39 SE; 3.93 SD)
Final number of mt haplotypes was 13.61 (0.22 SE; 2.15 SD)
Mean Ne calculated from loss of heterozygosity in extant populations: 877.50
移動のない場合
移動のない場合
Across all years, prior to carrying capacity truncation,
mean growth rate (r) was 0.0057 (0.0001 SE, 0.0172 SD)
Final expected heterozygosity was 0.8771 (0.0030 SE; 0.0299 SD)
Final observed heterozygosity was 0.8879 (0.0040 SE; 0.0403 SD)
Final number of alleles was 14.44 (0.22 SE; 2.21 SD)
Final number of mt haplotypes was 3.97 (0.11 SE; 1.11 SD)
Mean Ne calculated from loss of heterozygosity in extant populations: 238.79
男性のみが移動する場合において、わずかにmtDNAハプロタイプが減少しやすい傾向があるようで、男女をひっくりかえすと、「女性のみが移動する嫁入り婚形態では、Ychrの多様性が減少しやすい」という最初の仮説に合致します。ただ、まったく移動のない場合がトータルとしてはいちばんハプロタイプが減少しない、というのは私の想定外であり、まだまだ検証が必要なところです。
また、村ひとつで見ると移動がない場合は常染色体alleleもmtDNAのハプロタイプも大幅に減少することがわかります。
(2014/5/14追記)と書いてみたのはいいですが、男性のみが移動する場合、mtDNAの動向は移動のない場合とほとんど相似になるはず……ということはこの結果はやはり何かおかしいです。VortexでmtDNAがどのような扱いになっているのかいまひとつわかりません。
男性のみが移動する場合において、わずかにmtDNAハプロタイプが減少しやすい傾向があるようで、男女をひっくりかえすと、「女性のみが移動する嫁入り婚形態では、Ychrの多様性が減少しやすい」という最初の仮説に合致します。ただ、まったく移動のない場合がトータルとしてはいちばんハプロタイプが減少しない、というのは私の想定外であり、まだまだ検証が必要なところです。
また、村ひとつで見ると移動がない場合は常染色体alleleもmtDNAのハプロタイプも大幅に減少することがわかります。
(2014/5/14追記)と書いてみたのはいいですが、男性のみが移動する場合、mtDNAの動向は移動のない場合とほとんど相似になるはず……ということはこの結果はやはり何かおかしいです。VortexでmtDNAがどのような扱いになっているのかいまひとつわかりません。
PR
コメント
プロフィール
HN:
Haina
性別:
非公開
最新記事
(03/25)
(05/03)
(01/30)
(01/01)
(11/20)
